索引文件格式¶
在本节中,我们将使用“索引文件格式”,以纯文本配置为例
加载FASTA格式的索引¶
首先,我们假设正在建立Volvox mythicus(Volvox属中的一个神话物种)的基因组。 Volvox基因组已由测序中心在2018年进行了测序,他们想立即设置JBrowse。 他们为我们提供了指向其FASTA文件的链接,我们将下载该文件
mkdir data
curl -L https://jbrowse.org/code/JBrowse-1.16.7/docs/tutorial/data_files/volvox.fa > data/volvox.fa
我们将使用samtools的 faidx 命令来创建“ FASTA index”.
FASTA index能将非常大的FASTA文件“按需”下载到JBrowse中.
例如, 仅下载特定视图所需的序列.
samtools faidx data/volvox.fa
FASTA index将是一个名为volvox.fa.fai的文件。 然后,我们将这些文件移到JBrowse可以使用的“数据目录”中
然后创建文件data / tracks.conf,使用以下内容
[GENERAL]
refSeqs=volvox.fa.fai
[tracks.refseq]
urlTemplate=volvox.fa
storeClass=JBrowse/Store/SeqFeature/IndexedFasta
type=Sequence
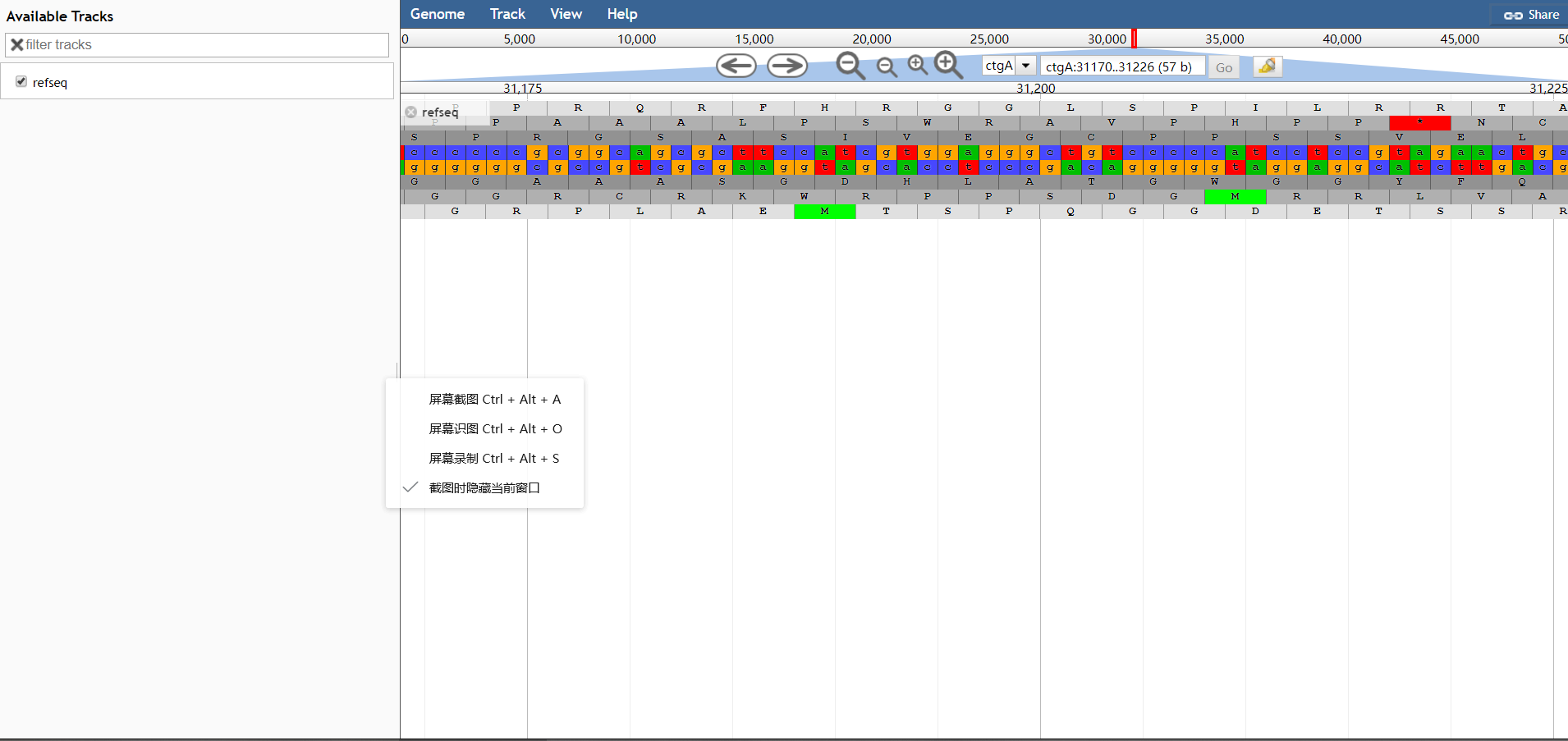
此时,您应该可以打开http://localhost/jbrowse/?data=data(或者打开http://localhost/jbrowse/), 您就会看到带有参考序列轨迹的基因组。 示例图如下:
加载Tabix GFF3¶
我们将使用的新生成的“基因注释”文件
curl -L https://jbrowse.org/code/JBrowse-1.16.7/docs/tutorial/data_files/volvox.gff3 > data/volvox.gff3
当我们处理GFF3以在JBrowse中使用时,我们的目标是使用GFF3Tabix格式。 T abix格式允许随机访问类似于Indexed FASTA的基因组区域。因此, 我们必须首先对GFF进行排序以为Tabx做准备
sort -k1,1 -k4,4n data/volvox.gff3 > data/volvox.sorted.gff3
bgzip data/volvox.sorted.gff3
tabix -p gff data/volvox.sorted.gff3.gz
在data/tracks.conf中加入一下配置:
[tracks.genes]
urlTemplate=volvox.sorted.gff3.gz
storeClass=JBrowse/Store/SeqFeature/GFF3Tabix
type=CanvasFeatures
完成!
加载BAM¶
如果已获得序列比对,也可以创建一个显示比对的“比对”轨迹 对于volvox,我们得到一个文件
curl -L https://jbrowse.org/code/JBrowse-1.16.7/docs/tutorial/data_files/volvox-sorted.bam > data/volvox-sorted.bam
请注意,此BAM文件已经排序。 如果您的BAM未排序,则必须对其进行排序以在JBrowse中使用。使用以下命令行:
samtools index data/volvox-sorted.bam
最后在data/tracks.conf中添加以下内容:
[tracks.alignments]
urlTemplate=volvox-sorted.bam
storeClass=JBrowse/Store/SeqFeature/BAM
type=Alignments2
检查您的文件是否已经加载成功¶
此时,如果jbrowse文件位于您的Web服务器中,则应具有目录布局,例如
/var/www/html/jbrowse
/var/www/html/jbrowse/data
/var/www/html/jbrowse/data/volvox.fa
/var/www/html/jbrowse/data/volvox.fa.fai
/var/www/html/jbrowse/data/volvox.sorted.gff3.gz
/var/www/html/jbrowse/data/volvox.sorted.gff3.gz.tbi
/var/www/html/jbrowse/data/volvox-sorted.bam
/var/www/html/jbrowse/data/volvox-sorted.bam.bai
/var/www/html/jbrowse/data/tracks.conf
你的tracks.conf文件夹中的内容如下:
[GENERAL]
refSeqs=volvox.fa.fai
[tracks.refseq]
urlTemplate=volvox.fa
storeClass=JBrowse/Store/SeqFeature/IndexedFasta
type=Sequence
[tracks.genes]
urlTemplate=volvox.sorted.gff3.gz
storeClass=JBrowse/Store/SeqFeature/GFF3Tabix
type=CanvasFeatures
[tracks.alignments]
urlTemplate=volvox-sorted.bam
storeClass=JBrowse/Store/SeqFeature/BAM
type=Alignments2
然后,您可以访问http:// localhost / jbrowse /将自动加载“data”目录。
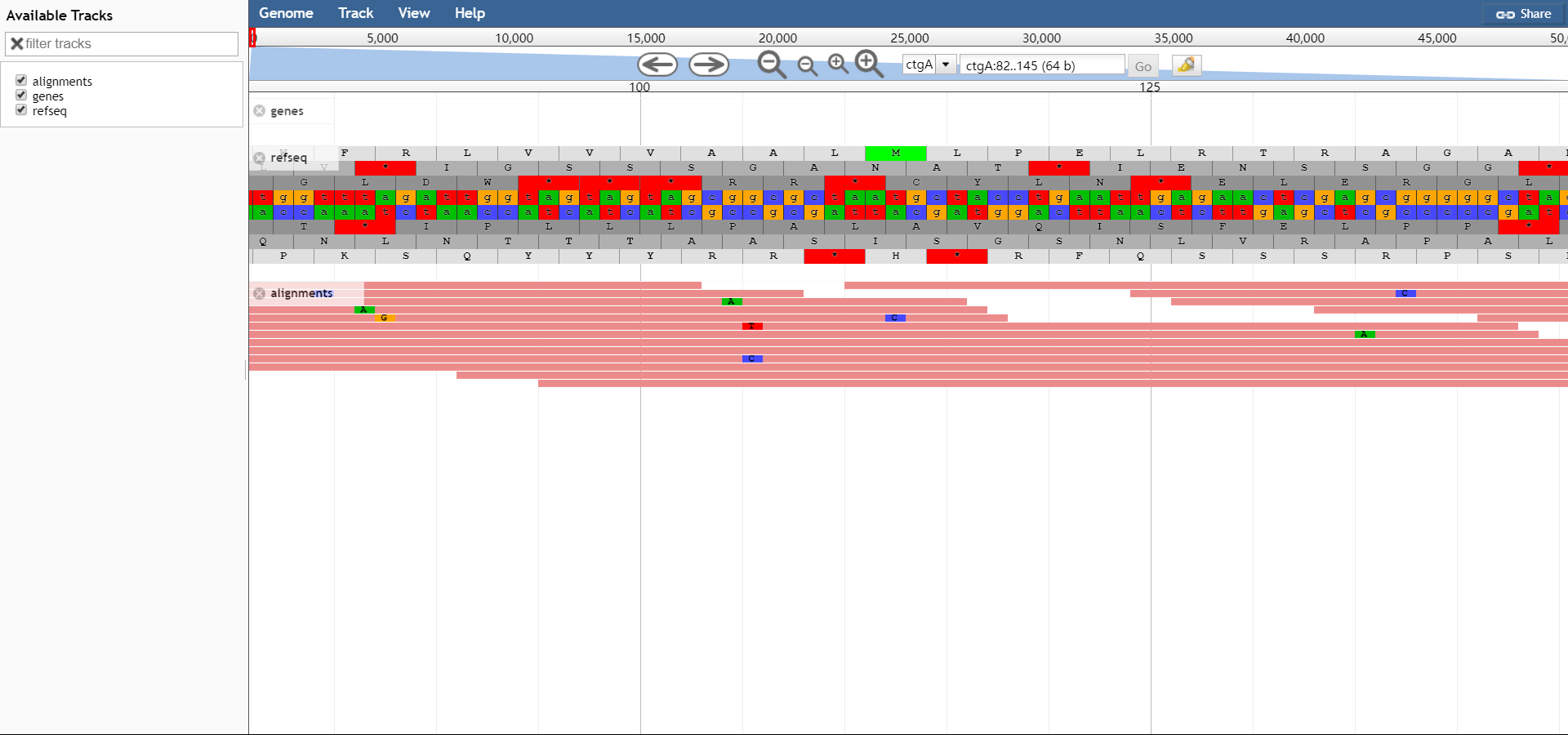
示例图如下:
祝贺!¶
您现在成功配置了JBrowse.